MetDNA - PN-III-P4-ID-PCE-2020-1292
Rezultate 2023
În a treia etapă a proiectului s-a urmărit analiza Raman ultrasenzitivă (SERS) a ADN-ului din trei linii celulare și clasificarea acestora pe baza analizei de tip machine learning (ML) a spectrelor SERS. Au fost înregistrate spectre SERS ale ADN-ului genomic (gADN) extras din celule control (linie ceulară HaCaT), ADN specific leucemiei acute mieloide (AML) (linie celulară NB4) și IDH mutant (linie celulară TF-1 cu mutația IDH). Pe baza scorurilor componentelor principale (PC) s-au construit modele de clasificare de tip machine learning: Naive Bayes, Random Forest, Logistic Regression, Support Vector Machine și kNN. Modelele au livrat acurateți de clasificare pentru cele trei linii celulare cuprinse între 70.4-92.6%, cu o tendință către o specificitate mai ridicată (76.9-97.9%) comparativ cu valorile pentru sensibilitate (75.5-92.6%).
Protocolul a fost repetat pentru probe de ADN extrase din probe de sânge de la subiecţi control, pacienți AML și pacienți cu mutație IDH. S-a urmărit astfel detecția AML pe baza analizei SERS a probelor de ADN extrase din sânge. S-a utilizat protocolul de detecție SERS optimizat pentru ADN genomic din etapele anterioare. Spectrele SERS achiziționate, utilizând protocolul optimizat în etapa 2, au determinat clasificarea probelor control, AML și IDH mutante cu acurateți între 72-90%. Rezultatele de clasificare au fost obținute folosind metode de clasificare (cross-validate) de tip machine learning: Naive Bayes, Random Forest, Logistic Regression, Support Vector Machine și kNN.
Tot în această etapă a proiectului s-a investigat prezența mutației IDH prin analiza SERS a plasmei sanguine. Activitățile au urmărit detecția oncometabolitului 2-hidroxiglutarat. Concentrația ridicată a acestui oncometabolit indică prezența mutației IDH la nivelul ADN-ului. În analiza probelor control, AML și IDH mutante a fost utilizat protocolul optimizat anterior pentru detecția și cuantificare a oncometabolitului 2HG din biofluide pe baza spectrelor SERS. Pe baza spectrelor au fost antrenate două metode de regresie de tip machine learning: Partial Least Squares (PLS) și Support Vector Machine (SVM), care au fost ulterior validate pe un set de probe. Factorul de corelare între concentrațiile reale de 2HG și cele prezise de modelul PLS a prezentat o valoare de corelare R2 de 0.702 cu o eroare medie pătratică de 0.007, iar pentru modelul de regresie SVM s-a obținut o corelație între concentrațiile reale de 2HG și cele prezise mai bună, cu un factor R2 0.956 cu o eroare medie pătratică 0.003.
De asemenea, proiectul s-a axat pe concentrarea selectivă a ADN-ului canceros în probe AML și IDH mutante. Pentru a arăta posibilitatea concentrării la suprafața nanoparticulelor a ADN-ului canceros, s-a marcat ADN-ul malign cu adenină marcată cu izotopul 13C. Activităţile efectuate au arătat că la un amestec de ADN malign/benign, cât suprafața de argint este mai mică, se observă mai bine banda caracteristică ADN-ului malign (la 709 cm-1), arătând faptul că atunci când avem o suprfață mică de argint (8 mm2) intervine competitivitatea adsorbţiei dintre ADN malign și benign pe suprafața de argint, ADN-ul malign fiind preferențial adsorbit.
Pentru probe cu raport inițial de 1:10 ADN malign la ADN benign, folosind protocolul optimizat s-a reușit îmbogățirea raportului la 1:1. Această concentrare a ADN-ului s-a realizat folosind o suprafață de argint echivalentă a 8 mm2 și o cantitate totală de 200 ng de ADN.
Protocolul de îmbogățire a ADN-ului la suprafața nanoparticulelor a fost aplicat pe n=15 probe AML și n=15 probe IDH detectate ca fiind cancer-negative. După protocolul de îmbogățire, analiza PCR a detectat mutația specifică leucemiilor PML-RARa în sașe probe.
Act.3.1. Clasificare probelor control, AML și IDH-mutante pe baza amprentei de metilare globală din spectrele SERS ADN
- Înregistrarea spectrelor SERS ADN din probe control, AML și IDH mutante
- Analiza statistică multivariată pentru clasificarea probelor control, AML și IDH mutante pe baza spectrelor SERS
Au fost înregistrate spectre SERS ale ADN-ului genomic (gADN) extras din celule control (linie ceulară HaCaT), ADN specific AML (linie celulară NB4) și IDH mutant (linie celulară TF-1 cu mutația IDH). Figura 3.1.1 prezintă spectrele SERS medii ale ADN-ului extras din aceste probe, evidențiind diferența spectrală dintre cele trei tipuri de ADN.
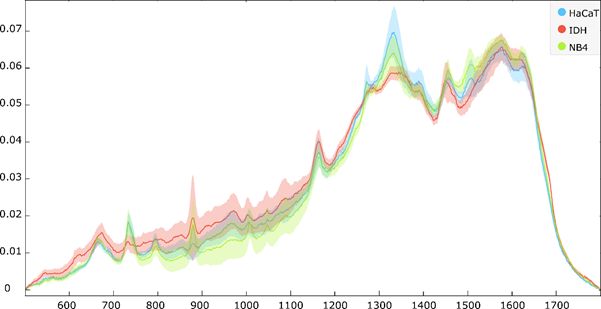
Figura 3.1.1.Spectrele SERS medii ale ADN-ului genomic extras din liniile celulare control (HaCaT), caracteristică leucemiilor (NB4) și linie celulară caracteristică leucemiilor cu mutație IDH (TF-1 cu mutația IDH). Spectrele sunt prezentate ca medie cu deviație standard.
S-a aplicat Analiza în Componente Principale (PCA) pentru a evalua prezența semnalului spectral discriminator pentru aceste trei tipuri de gADN. Score plot-ul rezultat din analiza PCA evidențiază posibilitatea utilizării spectrelor SERS ale ADN-ului din probe control, AML și IDH mutante în vederea clasificării originii ADN-ului (Figure 3.1.2).
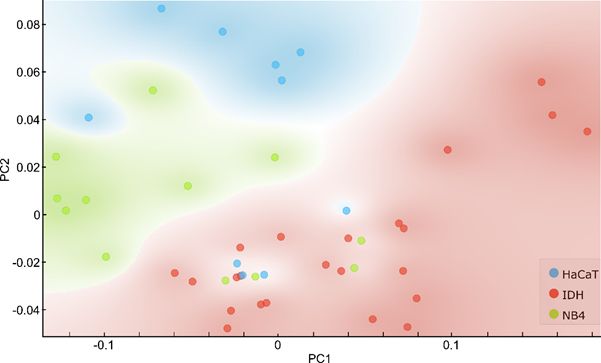
Figura 3.1.2. Score plot reprezentat de Componentele Principale 1 (PC1) și 2 (PC2) rezultat din analiza PCA aplicată asupra spectrelor SERS ale ADN-ului genomic extras din liniile celulare HaCaT, NB4 și TF-1 cu mutația IDH.
După reducerea dimensionalității datelor au fost păstrate 15 Componente Principale (PC) care exprimă 98.8% din varianța datelor. Pentru aceste PC-uri a fost analizată distribuția scorurilor în cele trei grupe de interes, control, AML, respectiv IDH prin utilizarea testului parametric neîmperecheat ANOVA. Au fost păstrate doar acele PC-uri pentru care testul ANOVA a arătat o diferență statistică în distribuția scorurilor pentru cele trei grupe, respectiv PC-urile pentru care valoarea p rezultată din testul ANOVA a fost mai mică de 0.05, respectiv PC1, PC2, PC3, PC5 și PC7.
Folosind scorurile acestor PC-uri s-au construit mai multe modele de clasificare de tip machine learning (Naive Bayes, Random Forest, Logistic Regression, Support Vector Machine și kNN) pentru clasificarea celor trei grupe. Pentru a evita overfitting-ul modelelor, acestea au fost cross-validate prin împărțirea setului de date în 80% grup training și 20% grup test. Procedura a fost repetată de trei ori, rezultatele fiind prezentate ca medie a celor trei evaluări.
Modelele de clasificare au livrat acurateți de clasificare cuprinse între 70.4-92.6 %, cu o tendință către o specificaitatea mai ridicată (76.9-97.9%) comparativ cu sensibilitatea (75.5-92.6%).
Același protocol a fost utilizat pentru probe de ADN extras din probe sanguine de la 25 pacienți control, 25 pacienți AML și 25 pacienți cu mutație IDH. Probele de ADN au fost extrase folosind un kit comercial (QIAamp DNA Mini Kit, QIAGEN). Metodologia de extracție adaptată pentru măsurătorile SERS dezvoltată pe studiul pe liniile celulare în etapa 2, a fost implementată în această etapă, probele de ADN fiind redispersate în apă ultrapură.
Cantitățiile de ADN extrase din aceste probe au variat între 46 ̶ 166 ng/µl pentru probele control, între 4 ̶ 236 ng/µl pentru probele provenite de la pacienți AML și între 10 ̶ 603 ng/µl pentru probele provenite de la pacienți cu mutația IDH. S-a observat o tendință către o cantitate mai mare de ADN în probele provenite de la pacienți cu cancer, însă această variabilă nu este relevantă statistic pentru clasificarea probelor.
Pentru aceste probe a fost determinată cantitatea de citozină metilată folosind metoda ELISA. Rezultatele au arătat o concentrație de citozină metilată între 0.16 ̶ 0.35 % pentru probele provenite de la pacienți AML și între 0.33 ̶ 0.80 % pentru probele provenite de la pacienți cu mutația IDH.
Act.3.2. Cuantificarea oncometabolitului 2HG prin SERS din probe reziduale control, AML și IDH mutante
- Înregistrarea spectrelor SERS a oncometabolitului 2HG din probe control, AML și IDH mutante
- Analiza statistică multivariată pe baza spectrelor SERS, pentru cuantificarea oncometabolitului 2HG din probe normale, AML și IDH mutante
Au fost înregistrate spectre SERS ale probelor de plasmă de la n=25 pacienți Control, n=25 pacienți AML și n=25 pacienți AML cu mutația IDH folosind protocolul dezvoltat în etapa 2 pentru detecția oncometabolitului 2HG. Probele analizate pentru prezența oncometabolitului 2HG din plasmă, provin de la aceiași subiecți analizați în activitatea 3.1, unde a fost prevăzută clasificarea pacienților pe baza semnalului spectral SERS al gADN.
Spectrele SERS au fost procesate conform protocolului dezvoltat în etapa 2, respectiv a fost păstrată regiunea spectrală 500-1800 cm-1, a fost redus background-ul folosind Rubber-band și spectrele au fost normalizate la vector. Figura 3.2.2 prezintă spectrele SERS medii ale probelor de plasmă de la pacienți Control, AML și IDH.
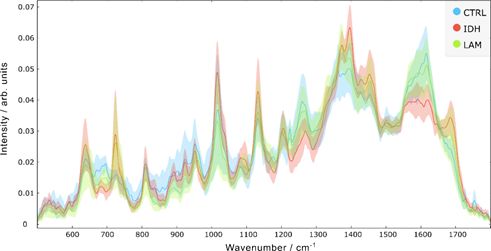
Figura 3.2.2. Spectrele SERS procesate ale probelor de plasmă de la n=25 subiecți control, n=25 pacienți cu leucemie acută mieloidă (AML) și n=25 pacienți cu AML cu mutație IDH. Spectrele sunt prezentate ca medie cu deviație standard.
În continuare, am construit două modele de regresie pentru cuantificarea concentrației de 2HG din probele de plasmă, Partial Least Square (PLS) respectiv Support Vector Machine (SVM). Modelele au fost construite folosind ca set de date de training 80% din setul de date inițial. Restul de 20% din date au fost folosite ca set de test pentru a valida modelelul de regresie de determinare a concentrației 2HG din probe folosind spectrescopia SERS. Au fost determinați hiperparametrii optimi acestor modele în determinarea concentrației de 2HG din probele de plasmă pentru cele trei grupe de pacienți control, AML și IDH. Pentru analiza PLS au fost determinați ca fiind optimi următorii hiperparametrii: 4 Componente cu 50 de iterații, iar pentru SVM au fost utilizți hiperparametrii Regression Cost C=2 și complexity bound v=0.9 folosind un Kernel de tip polinom de ordin 3.
Folosind hiperparametrii optimi determinați am testat cele două modele PLS și SVM pe setul de date test. Modelul PLS a rezultat într-un factor de corelare între concentrațiile reale de 2HG și cele prezise de model cu R2 de 0.702 cu o eroare medie pătratică de 0.007. Pe de altă parte, modelul de regresie SVM a obținut o corelație între concentrațiile reale de 2HG și cele prezise de model mai bună cu un factor R2 de 0.956 cu o eroare medie pătratică de 0.003.
Erorile de cuantificare a concentrației de 2HG din probele analizate sunt similare, indiferent de grupa control sau pacienți. Pentru modelul PLS avem o medie a erorilor de prezicere de 0.0033 pentru pacienții control fără 2HG, iar pentru modelul SVM de 0.0020. În cazul probelor ce conțin 2HG am obținut o medie a erorilor de prezicere similară de 0.0043 în cazul PLS, respectiv 0.0025 pentru SVM. Așadar nu se observă o tendință către o anumită clasă în erorile de prezicere.
Act.3.3. Concentrarea selectivă a ADN-ului canceros în probe determinate cancer-negative, datorită concentrațiilor sub limita de detecție
- Concentrarea ADN-ului canceros pentru 15 probe AML
- Concentrarea ADN-ului canceros pentru 15 probe IDH mutante
Pentru a putea urmări adsorbția selectivă a ADN-ului benign, respectiv a ADN-ului malign la suprafața nanoparticulelor ADN-ul canceros a fost etichetat cu adenină marcată cu izotopul C13 (Ade-C13) pentru 15 probe AML și 15 probe IDH mutante. Pentru aceasta liniile celulare AML au fost aduse în stadiul G0 și înfometate. Ulterior, în mediul de cultură s-a adăugat deoxidenozină marcată cu izotopul C13, care a fost consumată de liniile celulare și introdusă în genomul acestora pe parcursul a două zile. După cum se observă în Figura 3.3.1 ADN-ul a fost marcat cu succes cu Ade-C13, banda specifică adeninei de la 730 cm-1 fiind deplasată la 706 cm-1. Pentru a ne asigura că această modificare provine de la Ade-C13, am achiziționat spectre SERS pentru o probă de adenină, respectiv o probă de Ade-C13. Aceeași modificare a vibrației de la 730 cm-1 către 706 cm-1 a fost observată și în acest caz.
Pentru a concentra ADN-ul malign la suprafața nanoparticulelor de argint (AgNPs), a fost testată concentrația de ADN la care suprafața nanoparticulelor de argint este saturată cu ADN. Acest pas a fost realizat prin păstrarea constantă a cantității de nanoparticule și creșterea concentrației de gADN. A fost urmărită banda SERS specifică adeninei de la 730 cm-1, care crește cu cantitatea de ADN aflată la suprafața nanoparticulelor. Pentru o cantitate de 5 µl AgNPs corespunzătoare unei suprafețe de argint de 8 mm2 se observă o saturare a suprafeței de argint la o cantitate mai mare de 20 ng de gADN (Figura 3.3.2). Suprafața de argint a fost calculată din măsurătorile de diametru hidrodinamic al AgNPs și măsurătorile Nanotracking analysis ce au determinat concentrația nanoparticulelor. Astfel, diametrul AgNPs are o valoare de 60±6 nm ce corespunde unei suprafețe de argint de 2826 nm2, considerând NP ca fiind sferice. Pentru volumul de 5 µl AgNPs concentrația de AgNPs a fost determinată a fi 0.3x1010 NPs/ml. Astfel, suprfața totală de argint implicată în acest experiment este de 8 mm2.
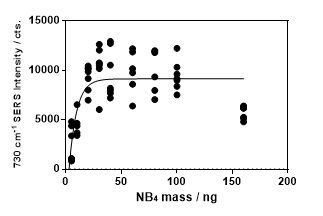
Figura 3.3.2. Curba de adsorbție a ADN-ului genomic pe suprafața de argint de 80 mm2 determinată în funcție de banda SERS caracteristică adeninei de la 730 cm-1.
Act.3.4. Măsurători PCR ale probelor concentrate selectiv
- Analiza PCR a ADN-ului canceros pentru 15 probe AML concentrate
- Analiza PCR a ADN-ului canceros pentru 15 probe IDH mutante
Pentru a analiza dacă semnalul multiplu prezent în analiza PCR provine de la fragmente de diferite dimensiuni a ADN-ului, toate probele ce au avut semnal specific genei PML-RARa la analiza PCR au fost testate prin electroforeză în gel. Figura 3.4.2. prezintă rezultatul migrării în gel de agaroză a celor 6 probe care au prezentat semnal la PCR. Nu s-a observat o diferență semnificativă între probele care au avut semnal dublu la analia PCR și probele care au arătat prezența genei PML-RARa, în ambele cazuri fiind prezente două dimensiuni de fragmente ADN.






